给分子育种带上精准“导航”
遗传变异是农业发展的基础,农作物育种的手段是创造和利用这些遗传变异。而分子育种就是利用分子生物学技术,将这些遗传变异进行组合(优良等位基因聚合)产生新的优良品种。目前随着高通量测序技术的发展,如GWAS、BSA、重测序等技术的应用,获得了丰富的遗传变异信息资源。在育种领域,这些遗传变异信息也可以称为分子标记(Molecular markers)。它们为分子育种提供了类似GPS导航的作用,使育种家可以清晰地了解不同株系或品种的性状特点和谱系,可以极大地加快育种进程,促进了下一代更优良品种的开发。利用这些分子标记,可以实现将功能基因组学的研究成果转变成大田作物优良品种的开发,从而创造巨大的经济效益,保障农业可持续发展的功能。
如何将分子标记低成本、大规模、自动化地应用于分子育种,给分子育种提供真正意义上的精准“导航”,让育种家们能够用的起,是近几年来一直研究的热点。真正实现低成本、大规模和自动化的应用,需要一种简单高效的分子标记检测技术。目前分子标记检测技术已经发展到第三代。其中,第一代是基于分子杂交技术的标记技术,如限制性片段长度多态性(Restriction fragment length polymorphism,RFLP);第二代是基于PCR技术的分子标记技术,包括序列标志位点(Sequence tagged sites,STS)、简单重复序列(Simplesequence repeat,SSR)、扩增片段长度多态性(Ampli-fied fragment length polymorphism,AFLP)等 ;第三代分子标记是基于对单苷酸多态性(Single nucleotide polymorphism,SNP)的分子检测技术。与第一、第二代分子标记相比,第三代分子标记SNP具有分布密度高、遗传稳定性好、二等位基因型等特点,更容易实现高通量和自动化检测。因此,SNP是目前分子标记技术中最适合大规模及低成本应用的。
基于SNP的分子检测技术,目前发展了出了几类高效、低成本的 SNP 基因型检测技术。第一类是基于竞争性等位基因特异性 PCR(Kompetitive allele-specific PCR)原理开发的高通量SNP检测技术,称为KASP技术。KASP技术由于其高通量、低成本和可操作性强等优点从而在农作物性状遗传和改良研究领域大量使用。该技术是基于引物末端碱基的特异匹配来对SNP位点进行检测,2个特异性结合引物在3’带有不同的等位基因识别位点,在第一轮 PCR 扩增中,等位基因特异引物(3’末端能配对的)就可识别特定等位基因模板,完成等位基因识别。从第2轮PCR扩增开始,产物中出现携带通用标签序列的模板,将把通用标签序列引入与SNP对应的PCR 产物中,在后面的PCR 扩增过程中,携带荧光的探针通过与通用序列互补的DNA链结合而添加到 PCR 产物中,经多轮PCR 扩增,更多的荧光探针退火到新合成的而逐渐增强 PCR 产物荧光强度。最后通过检测荧光信号而判别等位基因类型。其具体实验流程如下图1所示,其检测体系包括 :DNA模板、2个通用荧光探针、2个通用淬灭探针、2个与目标位点特异结合的引物以及共同的反向引物。该技术可以针对当个SNP位点进行大规模、自动化的检测,但是每次只能检测1个位点,应用场景受限。
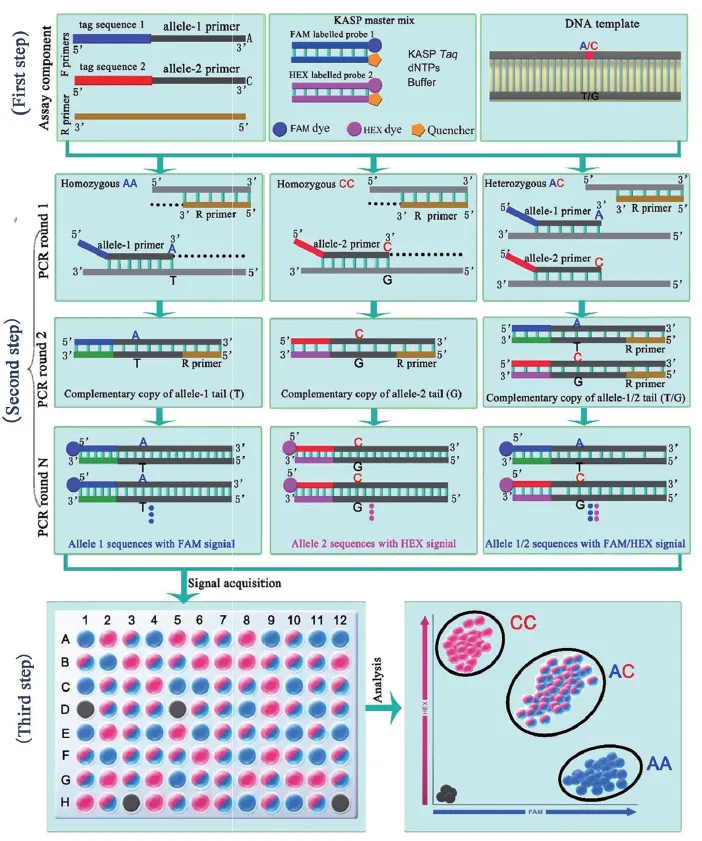
图1 KASP流程示意图(杨青青等,2022)。
第二类基于多重PCR技术的靶向捕获技术,对多个待测 SNP 位点设计特异扩增引物,在第一轮PCR中抑制引物干扰和非特异扩增,使数以千计的靶向特异性引物能够在一管PCR反应中实现均一化的扩增,从而大量富集目标片段。随后,在第二轮PCR中,加上测序接头和文库条形码,最终获得测序所需的文库,然后进行高通量测序,其实验流程如下图2所示。该技术可以实现单管高达2000对以上特定引物的混合扩增,一次检测可以实现对2000个位点的分析鉴定。该技术采用的多重PCR具有实验操作简单、成本低等特点,适合大规模、批量化的针对少于2000个位点以下的检测需求。
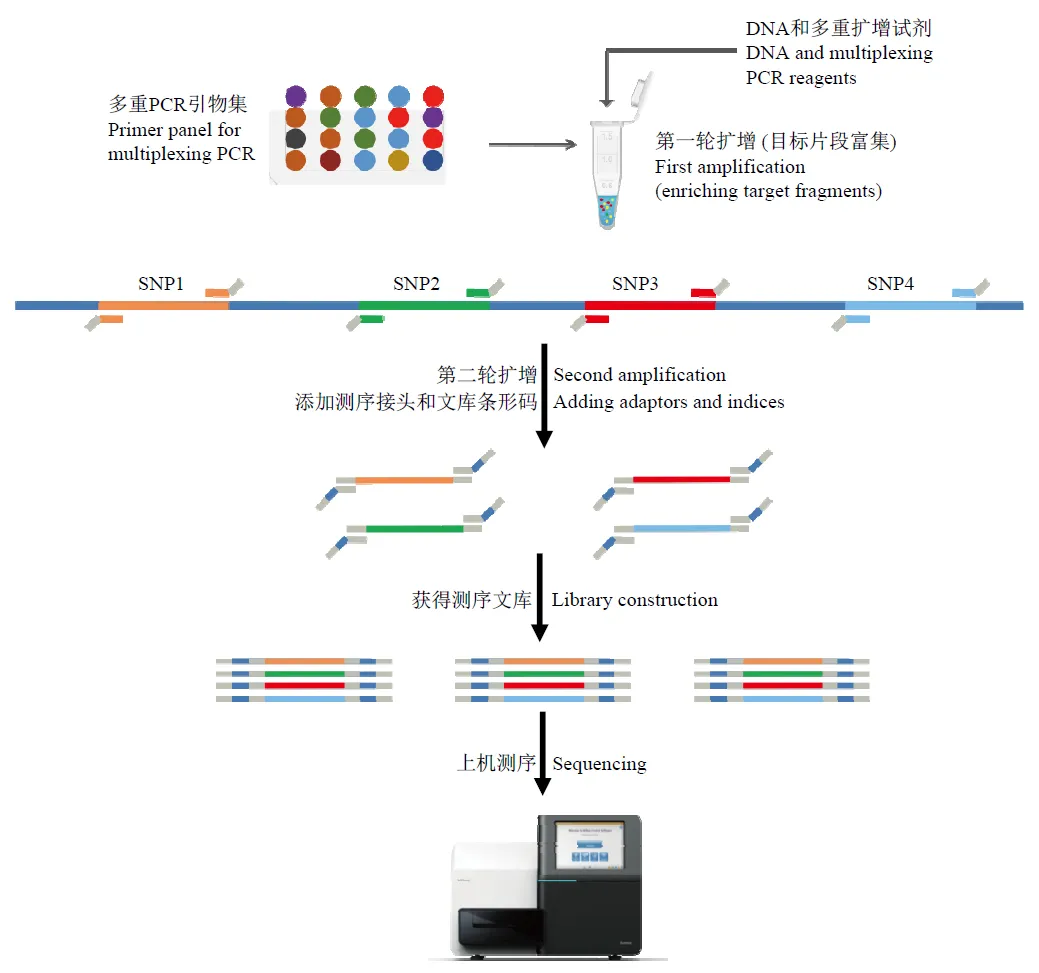
图2 基于多重PCR靶向测序的流程示意图(徐云碧等,2020)。
第三类液相探针靶向捕获测序技术,该技术采用目标探针与靶向序列互补结合进行定点捕获的特点,设计数以万计的探针,靶向捕获上万甚至数十万的的SNP进行测序分析。其实验流程如下图3所示,在每个待测位点设计覆盖目标SNP的探针,采用生物素(Biotin)标记对目标探针进行修饰。然后,在液态中利用生物素修饰的探针与基因组目标区域杂交形成双链。随后利用链霉亲和素包被的磁珠对携有生物素修饰的探针进行分子吸附,从而捕获与探针杂交的靶点。最后,对捕获的靶点序列进行洗脱、靶点扩增和测序,最终获得目标 SNP 的基因型。该技术可以对几十万个位点进行单管检测,而且单样本检测成本低。
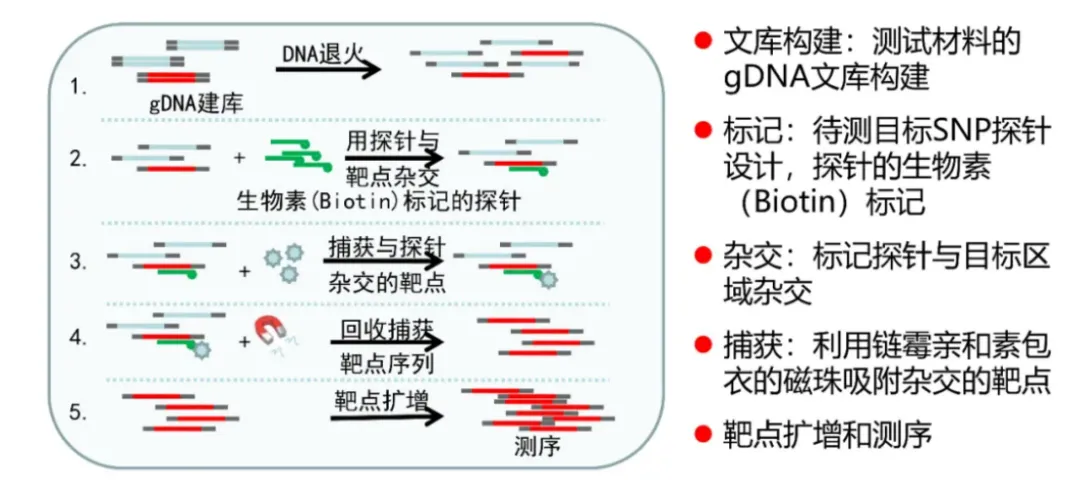
图3 基于液相探针靶向捕获技术的流程示意图。
在上述的技术方法的基础上,很多科研院校开发了不同的应用方法和技术。例如,江汉大学彭海教授团队利用多重 PCR结合二代测序开发的了MNP 法,并建立了实质性派生品种制度,明确了品种保护权的范围,为我国种业科技自强自立、种源自主可控提供坚实的法治保障。依托该方法,建立了《GB/T 38551-2020 植物品种鉴定 MNP 标记法》的法案,该法案是我国首个包括实质性派生品种判定的国家标准。

图4 GB/T 38551-2020植物品种鉴定MNP标记法。
北京市农林科学院蔬菜研究所温常龙团队利用多重PCR靶向捕获技术开发的Target SSR-Seq,使用122个SSR分子标记,对中国市场上382个主要黄瓜品种进行了基因分型,用来鉴别不同的黄瓜品种。

图5 基于多重PCR靶向技术鉴定黄瓜品系(Yang et al., 2019)。
田志喜研究团队利用液相芯片捕获技术,以2,898份材料的变异位点和等位基因频率为参考,选取了40,334个代表性的变异位点,设计开发了40K的大豆液相育种芯片,能够应用于大豆系统进化、群体遗传结构、全基因组关联分析(GWAS)、功能基因/QTL定位、分子设计育种相关表型预测等研究。
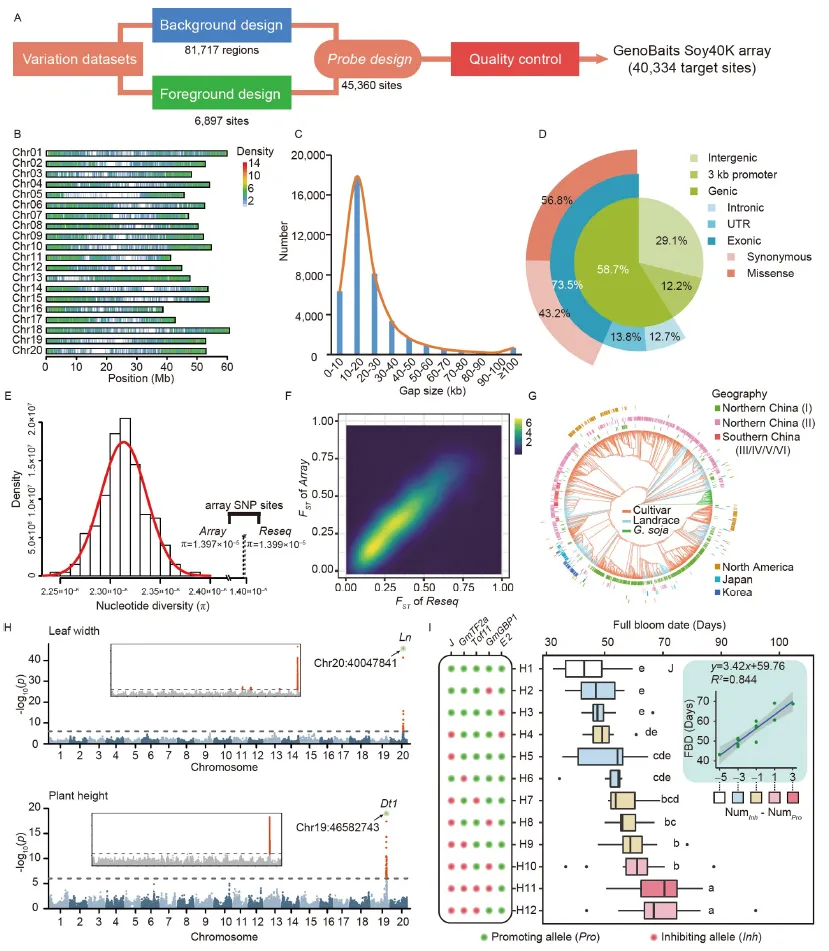
图6 基于液相靶向捕获技术开发的大豆40K液相育种芯片(Liu et al., 2022)。
这些技术的应用,为育种事业提供了一种低成本、高通量的检测方法,真正意义上为现在的分子育种事业加上了GPS导航,让育种家能够对候选品种的遗传背景了如指掌,加快育种事业的进程。
参考文献:
杨青青, 唐家琪, 张昌泉, 等. KASP 标记技术在主要农作物中的应用及展望[J]. 生物技术通报, 2022, 38(4): 58.
徐云碧, 杨泉女, 郑洪建, 等. 靶向测序基因型检测 (GBTS) 技术及其应用[J]. 中国农业科学, 2020, 53(15): 2983-3004.
Liu Y, Liu S, Zhang Z, et al. GenoBaits Soy40K: a highly flexible and low-cost SNP array for soybean studies[J]. Sci China Life Sci, 2022, 65(9): 1898-1901.
Yang J, Zhang J, Han R, et al. Target SSR-Seq: a novel SSR genotyping technology associate with perfect SSRs in genetic analysis of cucumber varieties[J]. Frontiers in Plant Science, 2019, 10: 531.
(来源:伯远生物)
上一篇:普通育种和转基因育种有什么区别?
下一篇:技术科普|从基因组到育种策略



